 Página anterior Página anterior |   Voltar ao início do trabalho Voltar ao início do trabalho | Página seguinte  |
Foram estudados os dados dos pesos à desmama (P240) de 5.498 animais com idade de 180 a 300 dias e dos pesos aos 18 meses de idade (P550) de 4.585 animais com idade de 490 a 610 dias nascidos entre os anos de 1954 e 2000, exceto 1955, pertencentes ao rebanho de bovinos Canchim da Embrapa Pecuária Sudeste, localizada no município de São Carlos, SP. Utilizaram-se apenas os dados de animais criados em pastagens e com suplementação mineral. Mayores informações sobre as condições de alimentação e manejo dos animais podem ser obtidas em Mello et al. (2002).
Foram organizados diferentes arquivos de dados: 1) arquivo com todos os pesos reais à desmama e aos 18 meses de idade disponíveis; 2) arquivo com os pesos à desmama e aos 18 meses de idade de animais cujo conjunto dos resíduos, após o ajustamento para os efeitos fixos do pai, do grupo de contemporâneos (animais do mesmo sexo e nascidos no mesmo ano e mês), da idade do animal no momento da pesagem (efeito linear) e da idade da mãe ao parto (efeitos linear e quadrático) apresentava coeficientes de assimetria e curtose próximos de zero, ou seja, foram eliminados os pesos que distorciam a curva de distribuição dos erros; 3) arquivo com todos os pesos padronizados à desmama (240 dias) e aos 18 meses (550 dias) disponíveis; e 4) arquivo com os pesos padronizados à desmama e aos 18 meses e que apresentavam coeficientes de assimetria e curtose próximos de zero.
Para padronização dos dados de pesagens à idade de 240 dias (desmama), foi utilizado o ganho de peso diário do nascimento à desmama e, para padronização à idade de 550 dias (18 meses), o ganho de peso diário dos 12 aos 18 meses de idade.
Os arquivos foram compostos pelos dados de animais que apresentaram pelo menos um dos pesos considerados (P240 ou P550). Assim, as diferenças quanto ao número de medidas de P240 e de P550 podem ser atribuídas a morte, a descarte de animais ou a eventuais falhas no processo de pesagem.
Foram realizadas análises exploratórias dos dados utilizando-se procedimentos do SAS (2000). Com essas análises, obtiveram-se as médias e os desvios-padrão, as modas e as medianas, os valores mínimos e máximos e os coeficientes de assimetria e curtose. Também foram realizadas análises de variância para verificar se a padronização dos pesos e/ou a eliminação de alguns deles provocaram alteração significativa nas médias dos pesos à desmama (240 dias) e aos 18 meses (550 dias).
Análises bivariadas do peso à desmama e aos 18 meses de idade foram realizadas para obtenção dos componentes de (co)variância porque, no rebanho estudado, não ocorre seleção de animais até o momento da desmama. Assim, espera-se evitar o efeito da seleção sobre os componentes de (co)variâncias para o peso aos 18 meses. Utilizou-se um modelo animal com os efeitos aleatórios genéticos aditivos direto e materno e de ambiente materno permanente (não correlacionado), além do resíduo. Os efeitos fixos considerados foram os de grupo de contemporâneos e de idade da vaca ao parto como covariável em dias (efeitos linear e quadrático). Foram também utilizados modelos contendo ou não a idade do animal no dia da pesagem como covariável (efeito linear).
A nomenclatura dos arquivos de dados e dos modelos estatísticos utilizados foi definida como a seguir:
· SP e CP: para os arquivos com os pesos reais e padronizados, respectivamente;
· SA e CA: para os arquivos contendo todos os dados disponíveis para análise e para os arquivos cujos dados que estavam distorcendo a distribuição foram excluídos, respectivamente; e
· SC e CC: para os modelos sem e com, respectivamente, a idade do animal no momento da pesagem como covariável.
Exemplo: SP_SA_SC correspondeu ao arquivo com os pesos reais (SP) de todos os animais, sem a exclusão dos dados que distorciam a distribuição (SA), analisado por um modelo sem a idade do animal como covariável (SC).
O modelo geral utilizado pode ser definido da seguinte forma matricial:
y = Xb + Z1a + Z2 m + Z3 q + e
em que y = vetor de observações (número de observações = n x 1); X = matriz de incidência (n x número de efeitos fixos = f) relacionando os efeitos fixos do vetor b (f ´ 1) aos animais correspondentes em y ; Z1(n ´ n), Z2(n ´ n) e Z3 (n ´ número de vacas = nv) matrizes de incidência que relacionam os efeitos aleatórios genéticos aditivos direto e materno e de ambiente materno permanente dos vetores a (n ´ 1), m (n ´ 1) e q (nv ´ 1), respectivamente, aos animais correspondentes em y; e e = vetor de erros aleatórios (n ´ 1). As pressuposições de distribuição dos efeitos aleatórios foram:
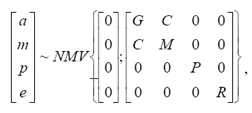
em que G = matriz de (co)variâncias genéticas aditivas diretas, G = As a 2; A = matriz de parentesco contendo 10.492 animais; M = matriz de (co)variâncias genéticas aditivas maternas, M = As m 2; C = matriz de covariâncias entre os efeitos genéticos aditivos diretos e maternos, C = As am ; P = matriz de (co)variância de ambiente materno permanente, P = Invs q 2; e R = matriz de (co)variâncias residuais, R = I s 2. ne.
As análises para obtenção dos componentes de (co)variância foram realizadas pelo método da Máxima Verossimilhança Restrita e os valores genéticos foram obtidos por meio do BLUP utilizando-se o programa MTDFREML (Boldman et al., 1993). A abordagem bayesiana para obtenção dos componentes de (co)variância e dos valores genéticos foi realizada por meio do Amostrador de Gibbs (AG) utilizando-se o programa MTGSAM (Van Tassel & Van Vleck, 1995). As análises pelo método da IB com AG foram realizadas utilizando-se cadeias de 330.000 ciclos, sendo que os 30.000 iniciais foram descartados e, então, foram obtidas amostras a cada 100 ciclos, totalizando 3.000 amostras dos componentes de (co)variância. Os hiperparâmetros que definem a forma da distribuição inicial dos componentes de (co)variância foram considerados iguais a zero. As amostras dos componentes de (co)variância obtidas pelo Amostrador de Gibbs foram ordenadas de forma crescente. Os intervalos das amostras 1 e 2.850, 2 e 2.851, ... e 151 e 3.000 foram calculados de modo que cada intervalo continha 95% das amostras (2.850). O menor intervalo foi escolhido para apresentação. Este intervalo pode ser denominado intervalo de alta densidade, pois representa o menor intervalo contendo 95% das amostras obtidas.
Após a estimação dos componentes de (co)variância e a predição dos valores genéticos, foi simulado um processo de seleção em que apenas 10% dos machos (25 touros) e 50% das fêmeas (836 vacas) com os maiores valores genéticos diretos para P240 e P550, para ambas as metodologias e os modelos estatísticos, permaneceriam no rebanho. Com base nas estimativas dos parâmetros obtidas e nos percentuais de seleção de machos e de fêmeas estabelecidos, foram calculados os ganhos genéticos esperados por geração ( DG ), por meio de: DG = had ´ S , em que: had representa a herdabilidade direta; e S corresponde ao diferencial de seleção. Neste caso: S = 1 (Sm + Sf ) , com Sm =im ´s p e Sf = if ´s p , em 2 que: im , if e sp representam as intensidades de seleção para machos e para fêmeas e o desvio-padrão fenotípico, respectivamente, de acordo com Pereira (2004).
Na Tabela 1 são apresentadas as estatísticas descritivas para os pesos à desmama e aos 18 meses de idade.
As médias dos pesos à desmama (P240) e aos 18 meses de idade (P550) e os desvios-padrão das médias estão dentro do intervalo encontrado na literatura para esta raça (Mascioli et al., 1996; ABCCAN-Embrapa-Geneplus, 2002), sugerindo que os resultados deste estudo podem ser generalizados para outros rebanhos da raça Canchim.
Não houve efeito significativo (P=0,26) da padronização sobre as médias do peso à desmama, porém, a eliminação dos pesos que estavam provocando distorção na curva de distribuição dos dados ocasionou aumento significativo (P=0,0004) das médias. Esse resultado foi verificado somente porque os coeficientes de assimetria iniciais eram negativos; dessa forma, a maior parte dos animais eliminados possuía pesos baixos, acarretando o deslocamento da média para a direita.
Houve efeito significativo (P=0,03) da padronização sobre as médias dos pesos aos 18 meses de idade, provavelmente porque a média de idade dos animais no momento da pesagem aos 18 meses era de 546 ± 16 dias e, como a padronização foi realizada para 550 dias, a maioria dos animais teve acréscimo no seu peso. A eliminação dos pesos que estavam provocando distorção na curva de distribuição dos dados não teve efeito significativo (P=0,92) sobre as médias do peso aos 18 meses porque os coeficientes de assimetria iniciais eram próximos de zero e a eliminação de dados foi realizada tanto para os animais que possuíam pesos elevados como para os que possuíam pesos baixos, apenas para reduzir a curtose.
Segundo Teixeira & Albuquerque (2003), a idade do animal na data da pesagem influencia o ganho de peso diário do animal em determinado período. Em razão desse fato, as discussões serão apresentadas em comparação ao arquivo com os dados padronizados e o modelo com a idade do animal no momento da pesagem como covariável.
Na Tabela 2 são apresentadas as estimativas dos componentes de (co)variância e dos parâmetros genéticos para os pesos à desmama e aos 18 meses de idade obtidas pelo método REML.
Considerando-se a idade do animal no momento da pesagem como covariável (efeito linear) e a metodologia
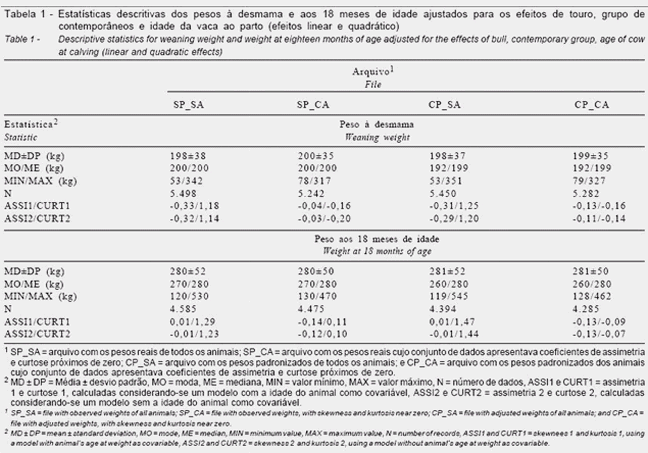
REML para estimação dos componentes de (co)variância, a análise do arquivo contendo os pesos reais dos animais cuja distribuição dos dados apresentava coeficientes de assimetria e curtose próximos de zero (SP_CA) foi a que forneceu as estimativas de (co)variância aditiva direta, herdabilidades e correlação mais próximas das obtidas quando foram estudados os pesos padronizados no arquivo em que os pesos de animais que estavam provocando distorção na curva de distribuição dos dados foram eliminados (CP_CA). É importante ressaltar que, nesses dois arquivos analisados, a padronização não provocou alteração significativa na média do peso à desmama nem no desvio-padrão e, mesmo existindo diferença significativa entre as médias reais e padronizadas do peso aos 18 meses, essa diferença foi inferior a 0,5% (Tabela 1), o que justica a pequena variação nos componentes de (co)variância.
Quando comparadas as estimativas de (co)variâncias aditivas e parâmetros genéticos obtidas nas análises do arquivo CP_CA às obtidas nas análises dos arquivos CP_SA e SP_SA, observou-se que as análises dos dois últimos forneceram estimativas de (co)variâncias aditivas superiores, porém, em virtude da maior variância residual, as herdabilidades foram inferiores às estimadas para o arquivo CP_CA. Este resultado corrobora os obtidos por Fonseca et al. (1999),que sugeriram que, quando a metodologia REML é utilizada para a estimação de componentes de (co)variância mas os resíduos não seguem distribuição normal, podem surgir desvios nas estimativas, contribuindo para o aumento na variância do erro de predição e diminuição dos ganhos genéticos. Dessa forma, a utilização de todos os dados disponíveis, sem a preocupação de excluir os dados que provocam distorções na distribuição dos resíduos, tenderia à escolha de indivíduos que não são geneticamente superiores e a prejuízos no progresso genético.
Ressalta-se que o teste de Kolmogorov-Smirnov (SAS, 2000) forneceu evidências de que a hipótese de que os resíduos seguem distribuição normal deve ser rejeitada, todavia, o procedimento de trabalhar com conjuntos de dados com coeficientes de assimetria e curtose próximos de zero tende a amenizar a situação de não-normalidade dos resíduos.
Quando a idade do animal no momento da pesagem não foi incluída no modelo, a análise do arquivo CP_CA forneceu as maiores estimativas de (co)variância aditiva direta e herdabilidades. Os componentes de (co)variância estimados nas análises dos arquivos CP_SA foram os mais próximos dos obtidos para o arquivo CP_CA, exceto para a herdabilidade do P550. As variâncias residuais obtidas na análise do arquivo CP_SA foram 40% superiores às obtidas para o arquivo CP_CA. O incremento da variância residual obtido na análise do arquivo contendo todos os dados disponíveis pode ser um indicativo de que a utilização do mesmo modelo estatístico para análise dos dois arquivos pode não ser a melhor alternativa. Por outro lado, a exclusão dos dados dos animais com baixos, ou com altos pesos, também deve ser cuidadosa, pois esses animais podem, realmente, ser de baixo ou de alto potencial genético.
Ainda considerando o modelo sem a inclusão da idade do animal no momento da pesagem como covariável, os componentes de (co)variância e as herdabilidades estimados na análise do arquivo SP_CA foram pelo menos 25 e 17%, respectivamente, inferiores aos obtidos na análise do arquivo CP_CA e os componentes de (co)variância e herdabilidades estimados na análise do arquivo SP_SA foram pelo menos 9 e 29%, respectivamente, inferiores aos obtidos na análise do arquivo CP_CA. Estes resultados divergem dos obtidos por Garnero et al. (1998), que, estudando os pesos à desmama e aos 18 meses em bovinos da raça Nelore, reais ou padronizados para 240 e 550 dias de idade, respectivamente, sem considerar a idade do animal no dia da pesagem como covariável, encontraram variâncias aditivas diretas e residuais superiores para os pesos reais em relação aos pesos padronizados, entretanto, a herdabilidade do peso à desmama foi superior para o peso padronizado e a herdabilidade do peso aos 18 meses de idade foi superior para o peso real.
Nas análises do arquivo CP_CA, a exclusão do modelo da idade do animal no dia da pesagem provocou aumentos superiores a 18% tanto nos componentes de (co)variância como nas herdabilidades e na correlação genética. Para o arquivo SP_CA, houve redução dos componentes de (co)variância e da herdabilidade (máximo 9%), enquanto, para os arquivos SP_SA e CP_SA, as variações foram de, no máximo, 10%. Estes resultados diferem dos obtidos por Pita & Albuquerque (2001), que não encontraram diferenças nos componentes de variância estimados para o ganho de peso médio diário de suínos quando a idade do animal no final do período experimental foi excluída do modelo. Todavia, considerando que a idade do animal no momento da pesagem influencia o ganho de peso diário (Teixeira & Albuquerque, 2003) e, conseqüentemente, o peso padronizado, a utilização desta covariável permitiria a comparação dos animais em igualdade de condições. Assim, acredita-se que, mesmo após a padronização do peso para determinada idade, é necessário incluir a idade do animal no momento da pesagem no modelo estatístico.
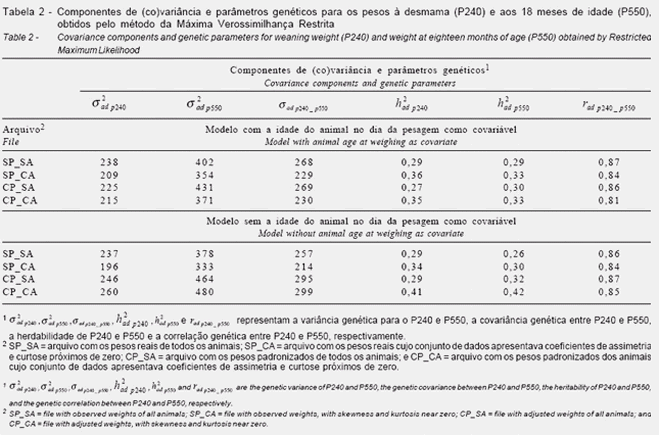
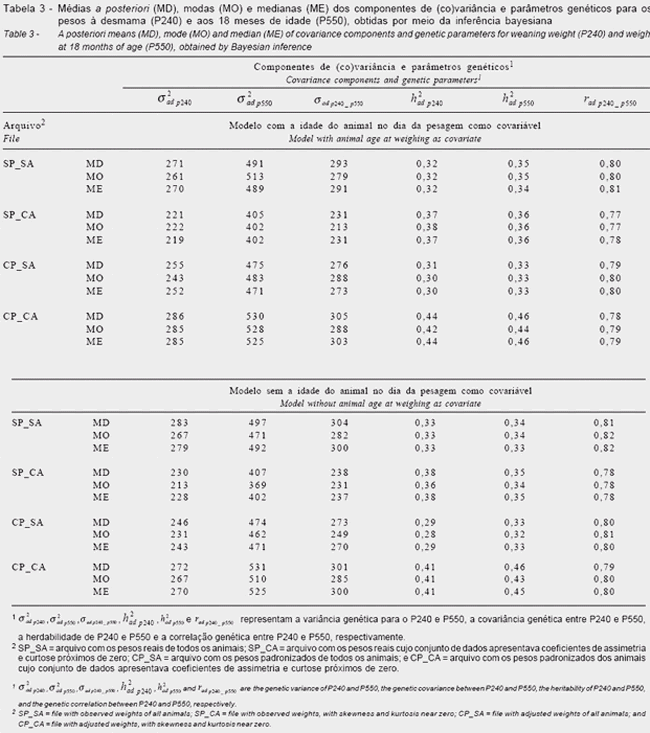
Na Tabela 3 são apresentadas as médias a posteriori, as modas e as medianas dos componentes de (co)variância e dos parâmetros genéticos para os pesos à desmama e aos 18 meses de idade obtidas por meio da inferência bayesiana.
Para os resultados obtidos utilizando-se a inferência bayesiana e o modelo com a idade do animal no dia da pesagem como covariável (efeito linear), as maiores médias a posteriori para os componentes de (co)variância e as herdabilidades foram obtidas na análise do arquivo CP_CA. As diferenças desses resultados com os encontrados na análise do arquivo SP_SA foram pequenas para os componentes de (co)variância (no máximo de 7%), mas, para as herdabilidades, foram maiores (24%), como conseqüência das maiores variâncias residuais obtidas na análise do arquivo SP_SA. A análise do arquivo SP_CA forneceu as menores médias a posteriori para os componentes de (co)variância (24% inferiores às obtidas na análise de CP_CA), no entanto, as diferenças para as herdabilidades foram menores (16 a 22%) em relação aos valores obtidos na análise do arquivo CP_CA. As diferenças entre as médias a posteriori para os componentes de (co)variância obtidas nas análises do arquivo CP_SA foram, em média, 10% e para as herdabilidades, 29% inferiores às obtidas na análise do arquivo CP_CA.
Quando a idade do animal não foi incluída como covariável no modelo, as médias a posteriori para os componentes de (co)variância obtidos nas análises dos arquivos CP_CA e SP_SA foram semelhantes, mas as herdabilidades foram distintas, sendo superiores nas análises do arquivo CP_CA (17% para P240 e 26% para P550). As médias a posteriori dos componentes de (co)variância e da herdabilidade de P240 obtidas nas análises dos arquivos SP_CA e CP_SA foram inferiores às encontradas nas análises dos arquivos CP_CA e SP_SA, sendo as médias a posteriori dos componentes de (co)variância obtidas na análise do arquivo SP_CA as menores.
Em todos os arquivos e modelos utilizados, as médias a posteriori foram superiores às estimativas obtidas por REML, exceto para as correlações genéticas, o que está de acordo com vários estudos da literatura utilizando dados de campo de bovinos (Magnabosco et al., 2001; Nobre et al., 2003a; Nobre et al., 2003b). Entretanto, com dados simulados, as médias a posteriori têm sido iguais às estimativas verificadas por REML (Van Tassell et al., 1995; Van Tassell & Van Vleck, 1996; Duangjinda et al., 2001). A conseqüência desse fato é que, quando os componentes de (co)variância são obtidos por REML (que, em geral, são inferiores aos obtidos por inferência bayesiana) e a seleção é baseada no BLUP, pode ocorrer tendência em atribuir maior importância.
às informações dos parentes dos reprodutores (Carneiro et a posteriori. Apesar das diferenças entre as médias al., 2001), levando à seleção de reprodutores com maior a posteriori e as estimativas obtidas por REML, a número de filhos. maioria dos componentes de (co)variância estimados por Na Tabela 4 são apresentados os intervalos de alta REML manteve-se dentro desses intervalos de alta densidensidade (95%) para as médias a posteriori para os dade para as médias a posteriori.
componentes de (co)variância e para os parâmetros gené-Neste aspecto, destaca-se uma vantagem da inferência ticos dos pesos à desmama e aos 18 meses de idade. Houve bayesiana: permite que as inferências sobre os parâmetros sobreposição dos intervalos obtidos para as médias em estudo sejam feitas com base nas distribuições

a posteriori, e não em um único valor, como na Máxima Verossimilhança Restrita (Patterson & Thompson, 1971; Van Tassell & Van Vleck, 1996).
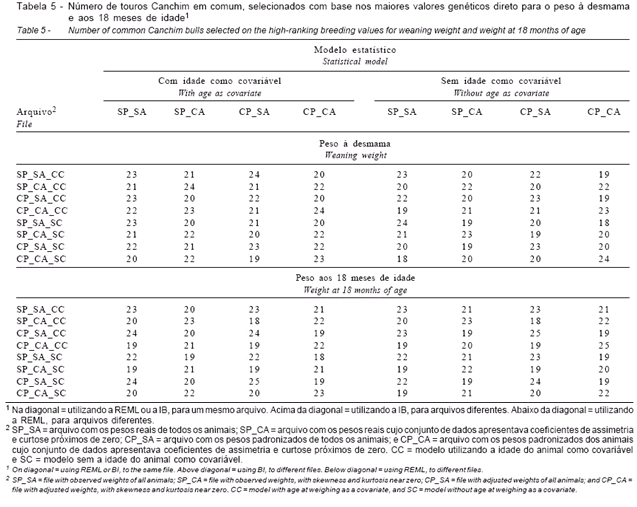
Na Tabela 5 são apresentados os números de touros em comum de um total de 25 selecionados com base nos maiores valores genéticos direto para os pesos à desmama e aos 18 meses.
Do total de touros (25) selecionados com base nos maiores valores genéticos aditivos direto para os pesos à desmama e aos 18 meses, apesar de terem ocorrido alterações nas classificações de posto, houve concordância em pelo menos 22 animais (88%), para um mesmo arquivo de dados, quando as avaliações genéticas foram realizadas por meio da REML ou da inferência bayesiana. Conseqüentemente, se os valores genéticos verdadeiros dos touros também fossem diferentes, poderia existir diferença na resposta à seleção massal. Quando utilizados arquivos diferentes, mas uma única metodologia, essas diferenças chegaram a 28% (18 animais em comum), como conseqüência dos componentes de (co)variância estimados, visto que, quanto mais semelhantes os componentes de (co)variância maior o número de animais selecionados em comum.
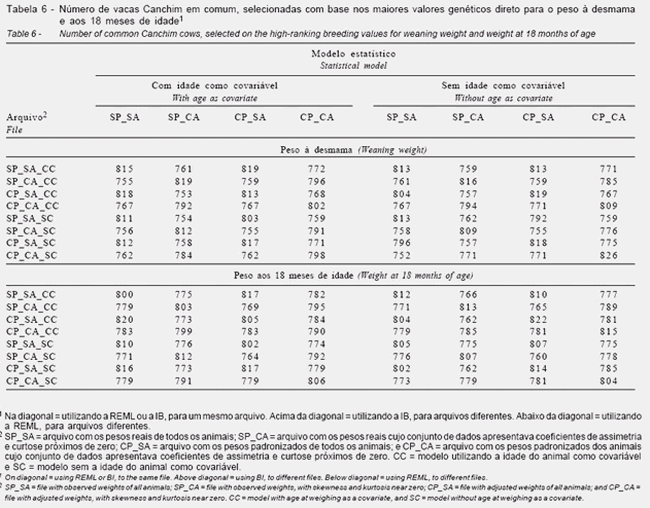
Na Tabela 6 são apresentados os números de vacas em comum de um total de 836 selecionadas com base nos maiores valores genéticos direto para os pesos à desmama e aos 18 meses.
Quando as avaliações foram conduzidas pela REML ou pela inferência bayesiana, pelo menos 96% (802 vacas) e 94% (790 vacas) dos animais foram selecionadas em comum, para um mesmo arquivo de dados, com base nos maiores valores genéticos aditivos direto para o peso à desmama e aos 18 meses, respectivamente, possivelmente em decorrência da menor intensidade de seleção para as vacas e da menor diferença entre o número de filhos de cada vaca, em relação aos touros.
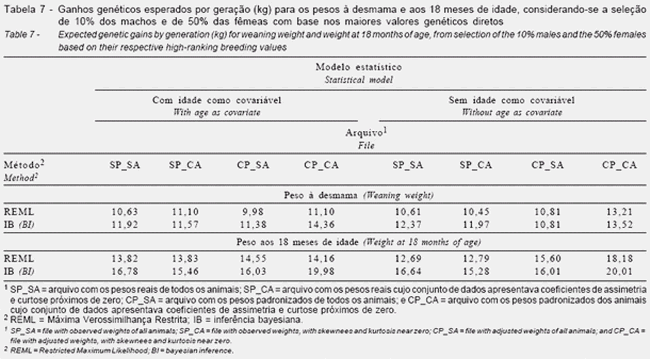
Constam na Tabela 7 os ganhos genéticos esperados e do modelo adotado, corroborando a possibilidade de por geração para os pesos à desmama e aos 18 meses de diferentes respostas à seleção. idade, considerando-se a seleção de 10% dos machos e de 50% das fêmeas e utilizando-se os componentes de variância Conclusões obtidos por meio dos métodos da Máxima Verossimilhança Restrita (REML) e da inferência bayesiana (IB).
Verificou-se (Tabela 7) que, apesar de os percentuais de touros e de vacas hipoteticamente selecionados em comum pela utilização das duas metodologias terem sido de 88% e de aproximadamente 95%, respectivamente, as diferenças no ganho genético esperado por geração variaram de 0 a 29% para o peso à desmama e de 2 a 41% para o peso aos 18 meses de idade, dependendo do arquivo analizado e do modelo adotado, corroborando a possibilidade de diferentes respostas à seleção.
No caso da avaliação de características de crescimento de bovinos da raça Canchim, como o peso, os resultados serão mais adequados se todos os animais forem comparados em mesma idade-padrão. Todavia, os efeitos que influenciam esta padronização ainda devem ser considerados. A utilização de diferentes metodologias para a avaliação genética animal, utilizando dados de campo, leva à identificação de diferentes animais como sendo genéticamente superiores. Dessa forma, podem ocorrer diferenças quanto ao progresso genético obtido nos rebanhos
ABCCAN-Embrapa-Geneplus. [2002]. Sumário de touros Canchim e MA. Edição Primavera/2002. Campo Grande: Geneplus Consultoria Agropecuária LTDA. Disponível em: <http://www.canchim.com.br/dep.htm#item7> Acesso em: 15/06/2003.
ALVES, R.G.O.; SILVA, L.O.C.; EUCLIDES FILHO, K. et al. Disseminação do melhoramento genético em bovinos de corte. Revista Brasileira de Zootecnia, v.28, n.6, p.1219-1225, 1999.
BOLDMAN, K.G.; KRIESE, L.A.; Van VLECK, L.D. A manual for use of MTDFREML. A set of programs to obtain estimates of variance and covariance (DRAFT). Lincoln: Department of Agriculture, Agricultural Research Service, 1993. 120p.
BLASCO, A. The Bayesian controversy in animal breeding. Journal of Animal Science, v.79, n.8, p.2023-2046, 2001.
CARNEIRO, A.P.S.; TORRES, R.A.; EUCLYDES, R.F. et al. Efeito da conexidade de dados sobre o valor fenotípico médio e a variância genética aditiva. Revista Brasileira de Zootecnia, v.30, n.2, p.336-341, 2001.
DUANGJINDA, M.; MISZTAL, I.; BERTRAND, J.K. et al. The empirical bias of estimates by restricted maximum likelihood, bayesian method, and method R under selection for additive, maternal, and dominance models. Journal of Animal Science, v.79, n.12, p.2991-2996, 2001.
FONSECA, R.; EUCLYDES, R.F.; TORRES, R.A. et al. Efeito da violação de pressuposições da metodologia de modelos mistos na avaliação genética animal. In: REUNIÃO ANUAL DA SOCIEDADE BRASILEIRA DE ZOOTECNIA, 36., 1999, Porto Alegre. Anais... Porto Alegre: SBZ/Videolar, [1999].
(CD-ROM) – Melhoramento Animal.
GARNERO, A.V.; LÔBO, R.B.; BORJAS, A.R. et al. Estimativas de parâmetros genéticos para características incluídas em critérios de seleção em gado de corte. In: REUNIÃO ANUAL DA SOCIEDADE BRASILEIRA DE ZOOTECNIA, 35., 1998, Botucatu. Anais... Botucatu: Sociedade Brasileira de Zootecnia,
1998. p.434-436.
HENDERSON, C.R. Selection index and expected genetic advance. In: HANSON, W.D.; ROBINSON, H.F. (Eds.) Statistical genetics and plant breeding. Washington, D.C.: NAS-NRC, 1963. p.141-153.
MAGNABOSCO, C.U.; FARIA, C.U.; BORJAS, A.R. et al. Implementação da amostragem de Gibbs para estimação de componentes de co-variância e parâmetros genéticos em dados de campo de bovinos Nelore. Planaltina: Embrapa Cerrados, 2001. 48p.
MASCIOLI, A.S.; ALENCAR, M.M.; BARBOSA, P.F. et al. Influência de fatores de meio sobre pesos de animais da raça Canchim. Revista da Sociedade Brasileira de Zootecnia, v.25, n.5, p.853-865, 1996.
MELLO, S.P.; ALENCAR M.M.; SILVA, L.O.C. et al. Estimativas de (co)variâncias e tendências genéticas para pesos em um rebanho Canchim. Revista Brasileira de Zootecnia, v.31, n.4, p.1707-1714, 2002.
NOBRE, P.R.C.; LOPES, P.S.; TORRES, R.A. et al. Analyses of growth curves of Nellore cattle by bayesian method via Gibbs sampling. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v.55, n.4, p.480-490, 2003a.
NOBRE, P.R.C.; MISZTAL, I.; TSURUTA, S. et al. Analyses of growth curves of Nellore cattle by multiple-trait and random regression models. Journal of Animal Science, v.81, n.4, p.918-926, 2003b.
PATTERSON, H.D.; THOMPSON, R. Recovery on inter-block information when block sizes are unequal. Biometrika, v.58, n.3, p.545-554, 1971.
PEREIRA, J.C.C. Melhoramento genético aplicado à produção animal. 4.ed. Belo Horizonte: FEPMVZ - Editora, 2004. 609p.
PITA, F.V.C.; ALBUQUERQUE, L.G. Efeitos da utilização de diferentes covariáveis na avaliação do ganho de peso médio diário em suínos. Revista Brasileira de Zootecnia, v.30, n.3, p.736-743, 2001.
STATISTICAL ANALYSIS SYSTEM - SAS. SAS user´s guide. Version 8.2. Cary: 2000. (CD-ROM).
TEIXEIRA, R.A.; ALBUQUERQUE, L.G. Efeitos ambientais que afetam o ganho de peso pré-desmama em animais Angus, Hereford, Nelore e mestiços Angus-Nelore e Hereford-Nelore. Revista Brasileira de Zootecnia, v.32, n.4, p.887-890, 2003.
Van TASSELL, C.P.; CASELLA, G.; POLLAK, E.J. Effects of selection on estimates of variance components using Gibbs sampling and restricted maximum likelihood. Journal of Dairy Science, v.78, n.3, p.678-692, 1995.
Van TASSELL, C.P.; Van VLECK, L.D. A manual for use of MTGSAM. A set of FORTRAN programs to apply Gibbs sampling to animal models for variance components estimation (DRAFT). Lincoln: Department of Agriculture, Agricultural Research Service, 1995. 86p.
Van TASSELL, C.P.; Van VLECK, L.D. Multiple-trait Gibbs sampler for animal models: flexible programs for Bayesian and likelihood-based (co)variance components inference. Journal of Animal Science, v.74, n.11, p.2586-2597, 1996.
Fábio Luiz Buranelo Toral1,
Maurício Mello de Alencar3,
Alfredo Ribeiro de Freitas3
1 Parte da dissertação de Mestrado do primeiro autor apresentada ao Programa de Pós-Graduação em Genética e Melhoramento Animal da Universidade Estadual Paulista/Faculdade de Ciências Agrárias e Veterinárias de Jaboticabal. Projeto financiado pela Embrapa e FAPESP.
2 Universidade Federal de Mato Grosso.
3 Embrapa Pecuária Sudeste. Bolsista do CNPq.
Recebido: 29/11/04
Aprovado: 04/07/06
Revista Brasileira de Zootecnia
© 2007 Sociedade Brasileira de Zootecnia
ISSN impresso: 1516-3598 R. Bras. Zootec., v.36, n.1, p.43-53, 2007ISSN on-line: 1806-9290
 Página anterior Página anterior |   Voltar ao início do trabalho Voltar ao início do trabalho | Página seguinte  |
|
|
|